こんにちは!はじめまして、カノンと申します。
それでは今日も細胞生物学のお話しをやっていきます。
今回のテーマはこちら!
動画はこちら↓
動画で使ったシートはこちら
組換えDNA技術原稿シート
それでは内容に入っていきます。
組換えDNA技術について
まず、組換えDNA技術とは、数百万個の塩基対のなかから特定のDNA断片を取り出し、複製し、元の生物や別の種類の生物の細胞に入れて機能させる技術のことです。
身近な例だと、大豆やトウモロコシ、ジャガイモなどを思い浮かべる方が多いかと思います。
厚生労働省のHPによると、遺伝子組換え食品は、安全性が確認された物だけが製造、輸入、販売される仕組みとなっており、安全性が確認された遺伝子組換え農産物とその加工食品については、食品衛生法及び JAS 法により表示が義務付けられています。
また、この組換えDNA技術は、PCR検査などの病気の判断や、核酸・抗体医薬などの治療にも大きな影響をもたらしています。
今回はこの技術の最も基本的な、目的のDNAを取り出して増幅し、コピーを多数作る過程についてご紹介したいと思います。
目的のDNAを取り出すには?
DNAの中には様々なタンパク質をコードする遺伝子が含まれています。
この大きなDNA分子から、ある目的のDNA配列を取り出すにはどうしたらよいのでしょうか。
それには制限酵素とよばれる、二本鎖DNAを特定の塩基配列のところで切断する酵素が必要になります。
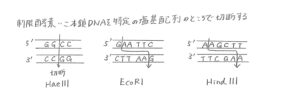
この制限酵素によって大きなDNA分子を小さな断片にし、ゲル電気泳動にかけることで、目的のDNA配列を取り出すことが可能になります。
詳しく見てみましょう。

網目構造をもったアガロースまたはポリアクリルアミドゲルにDNAを置き、電場をかけると負に荷電しているDNA断片は陽極に向かって移動します。
大きい断片ほどゲルの網目に引っ掛かりやすいのでゆっくり移動し、数時間後にはDNA断片は大きさに従って分離され、バンドがはしご状に並びます。
各バンドは大きさの等しいDNA分子の集まりなので、DNA断片を単離するにはゲルのその部分をカミソリで切り出して抽出します。
ただし、DNAのバンドはそのままでは目で見ることは出来ないので、DNAに結合するとUVによって蛍光を発する色素にゲルにさらしておきます。
こうすることで、ゲルをUV撮影装置で観察した際に、個々のバンドが確認でき、切り出しが可能になります。
このバンドは白黒写真で撮影することもできます。
組換えDNA分子の作り方
自然界では本来存在しない、「組換えDNA」の作製原理について簡単にお話します。
制限酵素とは、前述したとおり、二本鎖DNAを特定の塩基配列のところで切断する酵素であり、DNAリガーゼとは、細胞のDNAの複製や修復の際に生じるDNA主鎖の切れ目を連結する酵素のことです。
簡単に言うと、DNAを切ったり貼ったりすることのできる、「ハサミとのり」のような関係です。
また、プラスミドとは、小型の環状DNA分子のことで、細菌の細胞内で染色体と独立して複製するための複製起点のほか、外来DNA断片を挿入するための制限酵素切断部位を持っています。
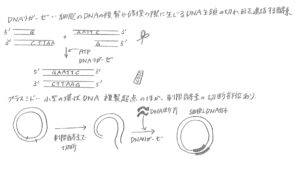
作製方法ですが、制限酵素でプラスミドDNAを切断し、DNAリガーゼを用いてDNA断片を挿入します。
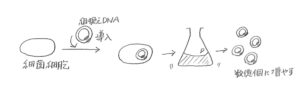
この組換えDNA分子を細菌に導入することで、複製され目的のDNA分子が増幅されます(およそ24時間ほどで目的のDNA断片をもつプラスミドが数億個できます)。
DNAにはイントロンとエキソンが存在します。
イントロンとは非翻訳領域であり、スプライシングによって取り除かれます。
エキソンは発現配列とよばれ、タンパク質をコードします。
ほとんどの真核生物では、エキソンはイントロンで分断されており、全体のDNAのほんの一部を占めていると言われています。
タンパク質をコードする遺伝子のクローニング(あるDNA塩基配列の複製を沢山つくること)には、翻訳されないイントロンの部分は必要ありません。
ではイントロンを全く含まないcDNAライブラリーの作成にはどうしたらよいのでしょうか。
mRNAを使用したcDNAライブラリーの作成
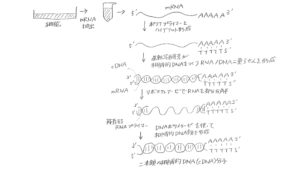
スプライシング後のmRNAを回収し、その配列をもとにcDNAをつくれば、ほとんどがタンパク質翻訳配列で、イントロンの部分が除去されたcDNAを作り出すことが可能です。
まず、全RNAを抽出し、逆転写酵素とDNAポリメラーゼによってRNA/DNAの二重らせんを形成させます。
その後、リボヌクレアーゼでRNAの部分分解を行い、残存するRNAプライマーを頼りにDNAポリメラーゼが再度相補的DNAの合成を進めます。
<用語補助>
ゲノムクローン:非翻訳領域DNA、イントロン、調節DNA、スペーサーDNAが大量に含まれている。その生物のどの細胞からとっても同じものができる。
cDNAクローン:ほとんどがタンパク質への翻訳配列であり、mRNAに転写された遺伝子のみ反映される。特定の細胞で、特定の時期、特定の条件下で発現する遺伝子を判断するのに利用される。
in vitroにおける目的のDNA断片の増幅
DNA二重らせんの2本の鎖は、相補的塩基対間の比較的弱い非共有結合(水素結合)をしており、90℃に加熱をすると2本の鎖を分離することが出来ます。
また、温度をゆっくりと下げると相補鎖同士が再度繋がり二重らせんを再形成します。
この性質を利用して、目的のDNA断片をin vitroで増幅することが出来ます。
増幅反応を何十回も繰り返す反復過程のことをPCR(ポリメラーゼ連鎖反応)といいます。
1回のPCRは下記の3段階からなります。
1.二本鎖DNAを短時間加熱して2本鎖を解離させる。
2.増幅したいDNA領域をはさむように設計した1対の特異的プライマーを加え、温度を下げる。すると、各プライマーはDNA鎖の相補的塩基配列とハイブリッドを形成する。
3.DNAポリメラーゼと4種類のデオキシリボヌクレオシド三リン酸を加え、各プライマーからDNAを合成させる。合成されたDNA鎖は再度熱処理によって解離させる。
1~3の一連の反応を繰り返す。

酵素は熱を加えると失活するイメージが強いと思いますが、DNAポリメラーゼは好熱性細菌のDNAポリメラーゼを使用しているので、熱処理しても変性しないようになっています。
まとめ
はい、今回の内容は以上です。
間違いの指摘、リクエスト、質問等あれば、Twitter(https://twitter.com/bakeneko_chem)
かお問い合わせフォームよりコメントしてくださると、助かります。
それではどうもありがとうございました!